论文分享|诺唯赞
论文分享▼
Cell Stem Cell
search
x
基本信息
x
Vazyme合作产品:
Hyperactive pA-Tn5 Transposase for CUT&Tag(Vazyme #S603)
Hyperactive Universal CUT&Tag Assay Kit for Illumina (Vazyme #TD903)
VAHTS mRNA-seq V3 Library Prep Kit for Illumina (Vazyme #NR611)
TruePrep DNA Library Prep Kit for Illumina (Vazyme #TD503)

x
研究背景
x
在人类基因组庞大的体系中,外显子只占了其中的1%-2%,其他非编码区域蕴含了大量的未知信息,这也是基因组的魅力所在。基因组内存在着可以移动的 DNA 片段——转座元件(Transposable Elements,TE),对于真核生物而言,除极少数物种外,几乎所有的真核物种基因组中都发现了TE的存在。人类基因组中有50%是TE,其中大部分是非长末端重复反转录转座子,例如SINE-VNTR-Alu(SVA)等元件,是基因组“暗物质”中的一种,对其深层功能的挖掘以及调控机制的研究将刷新我们对人类基因组的认知,同时也为人类胚胎发育内在机制的研究奠定基础。
x
论文概述
x
2022年7月7日,中山大学王继厂教授团队联合孙逸仙纪念医院王文军教授团队,在 Cell Stem Cell 期刊发表了题为“Dynamic reprogramming of H3K9me3 at hominoid-specific retrotransposons during human preimplantation development ”的研究论文。该论文探究了H3K9me3以及SVA元件如何参与人类早期胚胎发育,并解析了人胚胎细胞发育过程中H3K9me3特异性反转录转座子调节的动态变化对胚胎发育的重要影响(图1)。
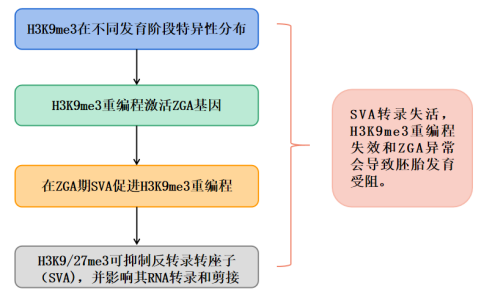
图1.技术路线
首先,由于传统的ChIP-seq技术需要百万级的细胞投入量,作者团队开发新的微量细胞ChIP-seq技术(AULiChIP-seq)打破了这一限制,仅投入50-500个早期人类胚胎干细胞就生成了高质量的H3K9me3 ChIP-seq数据(图2)。
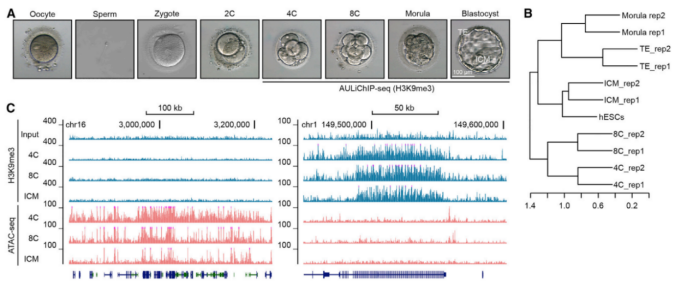
图2.人类植入前胚胎中H3K9me3的全基因组分析
(A)用于H3K9me3 AULiChIP seq实验的早期人类胚胎的代表性图像;
(B)人类早期胚胎和胚胎干细胞中全基因组H3K9me3信号的分级聚类;
(C)常染色质(左)和异染色质(右)的H3K9me3和ATAC-seq信号的代表性UCSC基因组图像。
其次,通过研究发现,在8细胞阶段,H3K9me3在反转录转座子SVA处重编程,促进了某些启动子和导向增强子之间的相互作用,促进了合子基因组的激活(ZGA)(图3),不论是SVA转录失活还是H3K9me3重编程失败都会阻碍ZGA和胚胎发育(图4)。
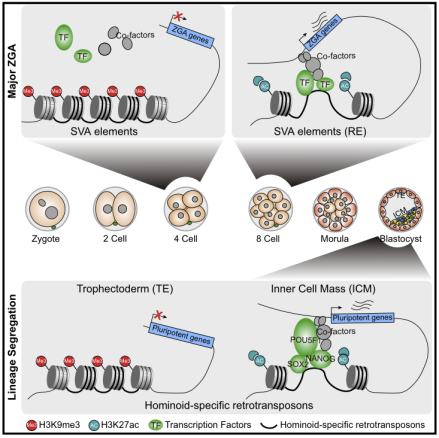
图3.H3K9me3和SVA对合子基因组激活基因和多能性基因的调控
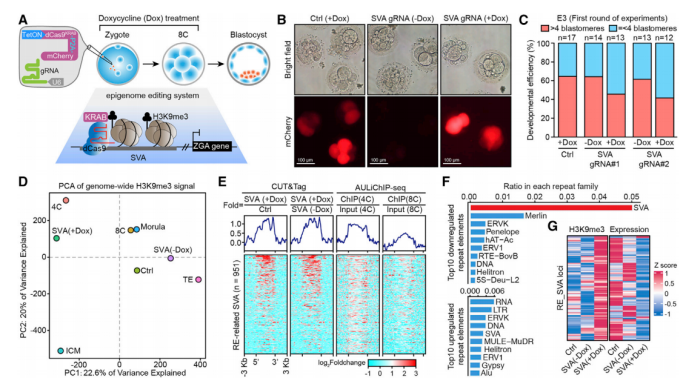
图4.H3K9me3在SVA位点的重编程失败阻碍人类ZGA和胚泡发育
(A)使用Dox诱导的dCas9KRAB系统在SVA位点维持H3K9me3依赖性异染色质的早期人类胚胎表观基因组编辑示意图;
(B)经表观基因组编辑的人类E3胚胎的图像;
(C)dCas9-KRAB介导的H3K9me3沉积的SVA抑制下人类E3胚胎的发育效率;
(D)人类野生型(WT)和表观基因组编辑胚胎中全基因组H3K9me3信号的主成分分析;
(E)通过CUT&Tag和AULiChIP-seq分析了人类4C和8C胚胎和表观基因组编辑胚胎中部分RE相关SVA位点的H3K9me3相对富集;
(F)与对照SVA(-Dox)胚胎相比,SVA(+Dox)胚胎中每个重复家族中top10上调或下调重复元件的比例。
最后,H3K9me3还在SVA上影响RNA的转录和剪接,SVA处的H3K9me3的状态可区分原始胚胎干细胞和内细胞团。总而言之,SVA转座子上的H3K9me3依赖性异染色质的物种特异性重组在人类早期胚胎发育过程中起着重要作用。
x
Vazyme产品支撑
x
在此次研究中,诺唯赞提供了CUT&Tag、RNA建库和DNA建库的产品支持。研究者使用了Vazyme #S603、Vazyme #TD903进行低起始量CUT&Tag实验(投入100个细胞),以及通过Vazyme #NR611和Vazyme #TD503进行RNA建库和DNA建库。

图5.诺唯赞产品文中应用(图片来自原文)
x
相关产品订购信息
x